現(xiàn)今��,科學(xué)界主流觀點認為線粒體起源于細胞內(nèi)共生古細菌����,因此線粒體DNA(mtDNA)與核基因組有著截然不同的進化軌跡【1】��。mtDNA位于線粒體基質(zhì)中,通過調(diào)控其拷貝數(shù)和表達水平來部分調(diào)節(jié)線粒體功能�,這種方式使線粒體能夠更好的響應(yīng)細胞內(nèi)能量需求和局部壓力【2】。mtDNA處于一個氧化壓力較大的線粒體微環(huán)境中����,加之mtDNA不與組蛋白結(jié)合�,缺乏核基因組的復(fù)雜調(diào)控網(wǎng)絡(luò),mtDNA的復(fù)制和轉(zhuǎn)錄過程較為寬松【3,4】�。核基因組的核苷酸修飾可以豐富DNA編碼的信息,使其超出四個典型堿基的范圍����,并在基因組調(diào)控中發(fā)揮重要作用。目前�,對于mtDNA是否具有功能性的核苷酸修飾仍是一個未有定論的猜想�。
近日�,來自澳大利亞昆士蘭大學(xué)的Steven Zuryn在Cell Metabolism上發(fā)表了論文Misregulation of mitochondrial 6mA promotes the propagation of mutant mtDNA and causes aging in C. elegans。在本研究中���,作者在多種真核生物mtDNA中鑒定到6mA的存在����,并揭示了6mA修飾在調(diào)控線粒體功能和機體壽命中的關(guān)鍵作用�����。

5mC修飾是脊椎動物核基因組中的主要表觀遺傳標記��,而6mA在原核生物中更為常見���??紤]到mtDNA的起源可能來自古細菌�����,因此對于mtDNA是否攜帶6mA修飾仍有爭論���。為了解決這個問題,研究人員利用多種方法去探究6mA修飾的存在����,包括了SMRT-seq、UPLC-MS�、MeDIP-qPCR和Dpn I甲基化敏感性檢測等。通過這些方法的綜合分析�����,作者在秀麗隱桿線蟲的mtDNA中證實了6mA修飾的存在,并且6mA修飾在多個物種中都可能存在���。這種修飾在不同物種中的高度保守可能反映了線粒體的原核起源假說。
接下來�,研究人員繼續(xù)尋找介導(dǎo)6mA修飾的候選酶。通過篩選����,最終鎖定甲基轉(zhuǎn)移酶DAMT-1部分定位于線粒體,特別是線粒體基質(zhì)和內(nèi)膜����。當DAMT-1缺失時���,mtDNA中的6mA水平低于對照組���,而將DAMT-1鎖定在線粒體中將大幅提高mtDNA的6mA水平����。此外,作者還發(fā)現(xiàn)SAM合成酶產(chǎn)生的S-腺苷甲硫氨酸對DAMT-1介導(dǎo)的mtDNA 6mA修飾至關(guān)重要。另一方面����,ALKB-1發(fā)揮了6mA去甲基化酶的作用,過表達ALKB-1將降低6mA的水平����。
在找到調(diào)控線蟲6mA修飾的甲基轉(zhuǎn)移酶和去甲基化酶后�,研究人員對6mA的生物學(xué)功能進行了研究。研究發(fā)現(xiàn)�,mtDNA的6mA修飾水平異常(無論是升高還是降低)都會顯著縮短線蟲的壽命,同時6mA修飾還調(diào)控了線粒體轉(zhuǎn)錄本的豐度��,并且在體細胞中限制了mtDNA的拷貝數(shù)��,6mA修飾的去除會導(dǎo)致mtDNA拷貝數(shù)的增加����。6mA對mtDNA的這些調(diào)控勢必會影響線粒體的功能��。的確��,作者進一步的研究發(fā)現(xiàn)�����,mtDNA 6mA的錯誤調(diào)控會引發(fā)線粒體與核基因組亞基之間的失衡,從而導(dǎo)致OXPHOS復(fù)合物組裝的缺陷�。這種缺陷不僅降低了線粒體的氧化磷酸化功能,還通過激活線粒體未折疊蛋白反應(yīng)(UPRmt)�����、增加氧化應(yīng)激水平等機制����,引發(fā)了與年齡相關(guān)的衰退和壽命縮短��。
文章的最后���,作者探討了mtDNA的6mA修飾對突變線粒體基因組的多代傳播的影響�����。研究結(jié)果表明���,mtDNA 6mA去甲基化通過增加mtDNA拷貝數(shù)�,促進了有害線粒體基因組突變在后代中的傳播和累積。這意味著mtDNA的6mA表觀遺傳狀態(tài)在跨代的mtDNA突變水平調(diào)控中起到了關(guān)鍵作用���。由于線粒體DNA通過母系遺傳����,6mA水平的變化可能對后代的健康和衰老有重要影響���,尤其是對于那些攜帶有害mtDNA突變的個體�。
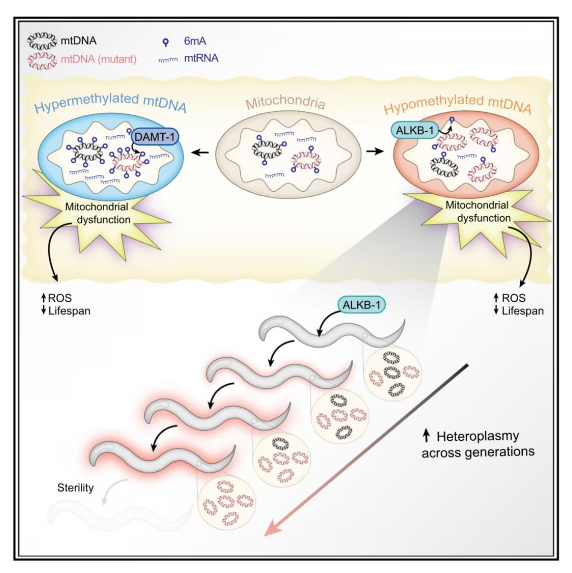
模式圖(Credit: Cell Metabolism)
總的來說�����,作者通過多種技術(shù)手段鑒定到6mA修飾在真核生物線粒體mtDNA中的存在��,并且篩選到關(guān)鍵的甲基化和去甲基化酶��,6mA修飾調(diào)控mtDNA的復(fù)制和轉(zhuǎn)錄以及突變的跨帶傳遞�����,最終影響線粒體功能和生物體壽命���。
參考文獻
1. Gray, M.W., Burger, G., and Lang, B.F. (1999). Mitochondrial evolution. Science 283, 1476–1481.
2. Johnston, I.G., and Williams, B.P. (2016). Evolutionary inference across eukaryotes identifies specific pressures favoring mitochondrial gene retention. Cell Syst. 2, 101–111.
3. Chinnery, P.F., and Samuels, D.C. (1999). Relaxed replication of mtDNA: A model with implications for the expression of disease. Am. J. Hum. Genet. 64, 1158–1165.
4. Sastre, J., Pallardo ? , F.V., and Vin ? a, J. (2003). The role of mitochondrial oxidative stress in aging. Free Radic. Biol. Med. 35, 1–8.
美鳳力借多年的大動物經(jīng)驗,堅持以“務(wù)實求真”為宗旨��,累計為4000多家客戶提供品質(zhì)大動物臨床前服務(wù)��,得到了客戶的一致好評��。如果您有動物試驗����、臨床培訓(xùn)�、組織病理、大動物試驗���、臨床試驗�����、產(chǎn)品注冊科研課題等...
請立即點擊咨詢我們或撥打咨詢熱線: 17312606166 ����,我們會詳細為你一一解答你心中的疑難��。 添加好友




 17312606166
17312606166